
Mai 2025 – Décrire les codes LOINC avec SNOMED CT
Date Article : 20 mai 2025
Type d’article : Regard d’expert – (L’équipe CGTS de l’ANS vous apporte une analyse sur un sujet d’actualité dans la cadre de sa veille scientifique & technologique)
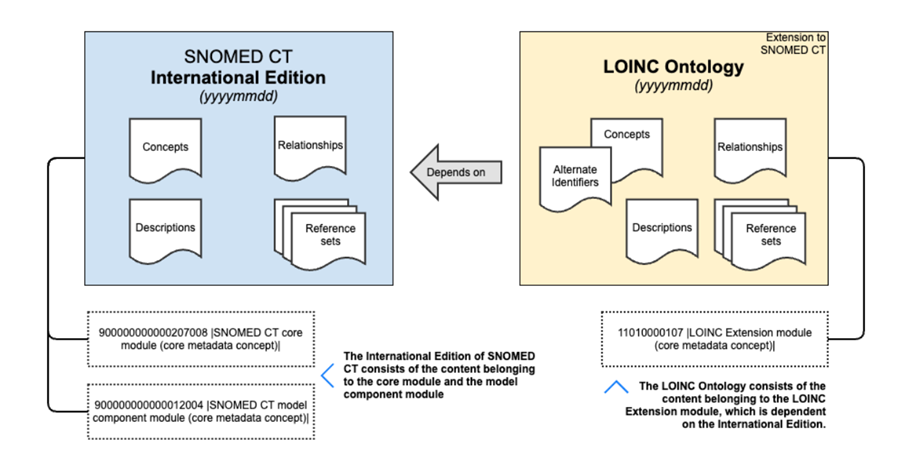
Depuis mars 2025 la version 2.80 de LOINC est accompagnée de la version 1.0 en production de l’ontologie publiée sur le site https://loincsnomed.org/. Cette version a été présentée le 25 mars lors d’un webinaire. Le projet de description formelle des codes LOINC sur la Terminologie SNOMED CT résulte d’un partenariat entre le Regenstrief Institute et SNOMED International, ravivé en 2022.
Cet article couvre certaines des informations dont nous disposons à l’ANS, et nous avons décortiqué tout cela et vous présentons ci-dessous notre analyse.
Ce projet permet de faire converger la description des codes LOINC dans le système de représentation des connaissance SNOMED CT pour améliorer l’intégration et l’exploitation des données patients dans les dossiers médicaux électroniques et entrepôts de données de santé. Si la première version de l’ontologie ne couvre que 35 000 codes LOINC de laboratoire, à terme l’objectif est bien de représenter tous les codes LOINC en SNOMED CT sans pour autant fusionner les deux Terminologies. En effet, l’ontologie LOINC est distribuée sous forme d’extension SNOMED CT et vient compléter les formats de distributions actuels de la Terminologie (téléchargeable sur le site http://loinc.org).
« By aligning these terminologies, the extension supports data harmonization, advanced analytics, and improved patient care. “
Guide utilisateurs LOINC-SNOMED CT
Couverture : les codes pour le Laboratoire
L’ontologie couvre 35 344 codes de laboratoire dont 75% du top 2000 des codes LOINC les plus utilisés aux USA. La figure ci-dessous présente le taux de couverture de de l’ontologie par discipline.
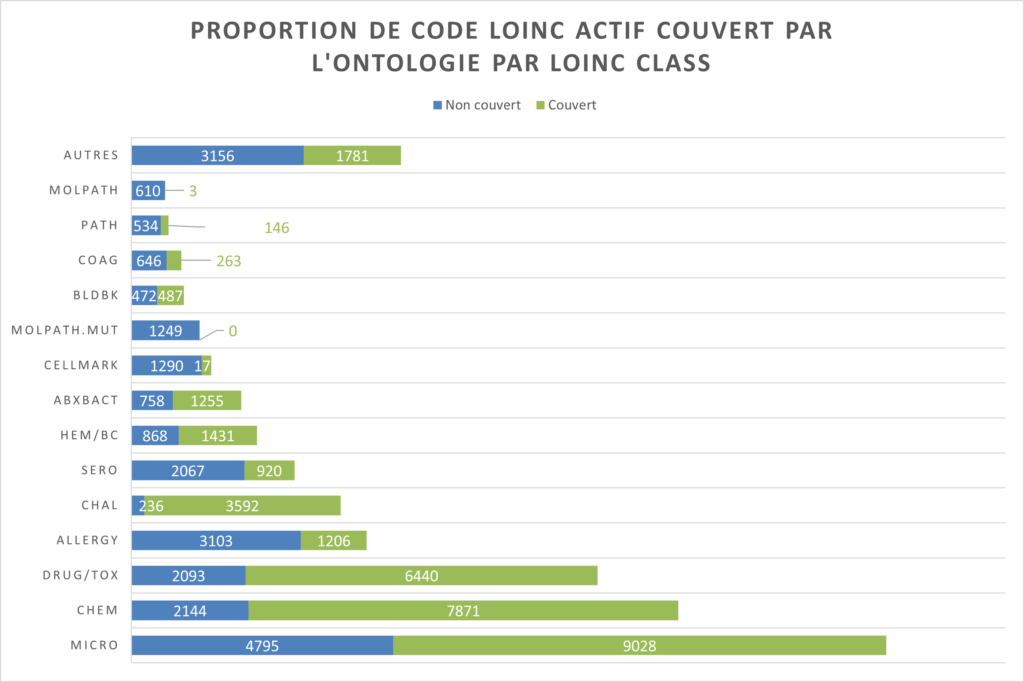
En comparaison, le taux de couverture des codes en usage en France est plus faible : seul 59% des codes analyses du Circuit de la Biologie1 et 9,4% des codes du top 200 des analyses les plus fréquemment prescrites (colonne Liste retreinte) sont dans cette version.
Modèle & requêtage
Les codes LOINC de laboratoire sont décrits dans la hiérarchie 363787002 |entité observable| qui représente des éléments (qualité, tendance, fonction, processus) à observer et la façon dont ils seront observés. Le projet a permis de revoir la modélisation des codes LOINC en fonction de l’objet du test (voir guide d’implémentation https://snomed.org/loinc).Le guide indique que 8 patrons de conception ont été définis en fonction de :
- si l’analyse mesure la présence ou quantité d’un analyte ;
- si l’analyse exprime un ratio entre deux analytes ;
- si le test correspond à une observation sur l’échantillon ;
- …
L’exemple ci-dessous montre les différences de modélisation pour deux codes LOINC permettant de mesurer la présence d’ADN de microorganisme par PCR. La relation décrivant l’analyte mesuré est différente en fonction de la nature du résultat :
- Le code LOINC 35691-5 utilise l’attribut Component pour exprimer la présence d’ADN alors que
- Le code LOINC 38347-1 permettant de caractériser l’ADN d’un microorganisme utilise l’attribut Inheres In


Ces nouveaux modèles permettent une description formelle et précise de la signification d’une analyse. Les règles d’implémentation documentées dans le guide éditorial sont garantes de leur reproductibilité mais apportent peu ou pas d’explication métier permettant de comprendre le sens des patrons. Si à première vue, la description plus complexe d’un code LOINC semble être un frein au requêtage de la Terminologie, c’est sans compter l’efficience des moteurs de recherche ECL pouvant être utilisé sur la SNOMED CT (http://snomed.org/ecl) .
L’ECL permet de générer des requêtes complexe en combinant :
- des opérateurs logiques AND et OR
- des opérateurs hiérarchiques permettant d’augmenter ou restreindre les concepts sélecteurs et résultats aux descendants directs (<!) ou à l’ensemble de la sous hiérarchie (< et <<)
- des opérateurs joker (*) permettant de ne pas spécifier un concept ou un attribut dans la requête
- des opérateurs de sélection de codes dans des reference sets, permettant de filtrer sur les codes LOINC Orderable (635111010000100 |LOINC Orderable Reference Set|) ou Observation (635121010000106 |LOINC Observation Reference Set|)
- des opérateurs de filtre textuel applicable aux libellé et définition littérales.
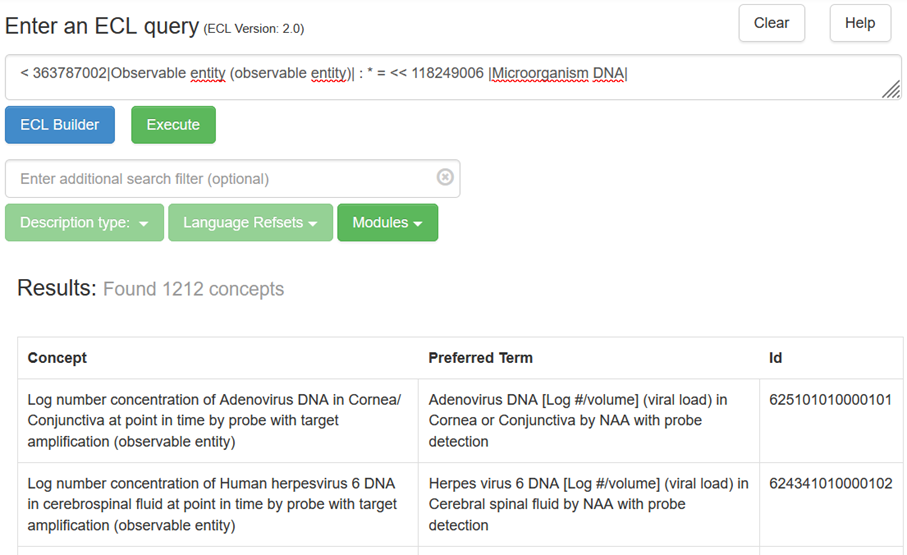
Ainsi il est par exemple possible de générer une requête agnostique du patron de conception en utilisant l’opérateur joker *, pour sélectionner par exemple tous les codes associés au concept 118249006 |Microorganism DNA| (exemple 1) ou étendus à lui et ses descendants (exemple 2).


Les bénéfices pour l’intégration et l’exploitation des données de Biologie
L’usage de la SNOMED CT offre de nouvelles perspectives sur la navigation et la recherche de codes LOINC.
Tout d’abord, la formalisation des codes LOINC avec des concepts SNOMED CT permet d’utiliser la polyhiérarchie SNOMED CT pour créer une arborescence de codes. Dans l’exemple ci-dessous le concept 38347-1| Presence or identity of microorganism DNA in specimen at point in time by probe with target amplification (observable entity) ne spécifie pas à quel microorganisme l’ADN testé appartient ni la nature de l’échantillon. L’arborescence présente des concepts fils plus spécifiques sur ces deux axes. En proposant une organisation hiérarchique, l’ontologie facilite la navigation dans la Terminologie LOINC en classifiant des concepts avec des définitions proches. Attention cependant à ne pas exploiter cette hiérarchie pour ré-interpréter des résultats d’analyse, cela peut amener des effets de bords. Par exemple, le résultat « absence d’ADN de Brucella dans un échantillon de sang » n’implique pas « l’absence d’ADN de microorganisme dans un échantillon non spécifié » (pour plus de détails voir l’article2).

Ensuite, la modélisation en graphe de la SNOMED CT couplé au langage ECL facilite la sélection de codes LOINC répondant à une logique métier par cas d’usage. Il devient par exemple possible de sélectionner les codes LOINC permettant d’identifier l’ensemble des microorganismes de type levure associés à des Infections Sexuellement Transmissibles. Pour réaliser la même requête dans une autre interface de recherche il aurait été nécessaire de lister de manière exhaustive les levure/champignons.

Cela ouvre des perspectives très intéressantes dans les cas d’usage d’exploitation de données et production de soin. En effet, l’ontologie LOINC couplée à SNOMED CT peut, par exemple, simplifier la constitution de cohorte en facilitant la sélection de tests orientés sur des pathologie. En production de soin les SI de laboratoire pourraient également bénéficier de cette ontologie pour, par exemple, faciliter la recherche de codes et générer des sous listes de résultats compatibles avec le code LOINC sélectionnés.
L’ontologie LOINC – une intégration dans SNOMED CT prometteuse
Pour conclure, l’ontologie LOINC 1.0 est prometteuse, son implémentation peut faciliter l’adoption de LOINC en apportant de nouveaux services aux utilisateurs (hiérarchie, requête). Néanmoins le taux de couverture actuelle n’est pas suffisant pour une implémentation nationale sur le cas d’usage de la Biologie.
Si d’aventure vous vous lancez dans une implémentation conjointe, n’hésitez pas à nous partager vos retours d’expérience !
- basé sur les analyses actives présent dans la version 1.1.22 ↩︎
- Mary, M., Soualmia, L. F., Gansel, X., Darmoni, S., Karlsson, D., & Schulz, S. (2017). Ontological Representation of Laboratory Test Observables: challenges and perspectives in the SNOMED CT observable entity model adoption. In Artificial Intelligence in Medicine: 16th Conference on Artificial Intelligence in Medicine, AIME 2017, Vienna, Austria, June 21-24, 2017, Proceedings 16 (pp. 14-23). Springer International Publishing. ↩︎